午前
・標本整理
午後
・論文書き(マダニ卒論論文化)
解析のスピードは結構改善されたと思っていたが、英語を書くスピードは相変わらず遅い。
これを片付けて、今年はマダニから離れてワラジムシ類に集中した。
2020年3月30日月曜日
anti_join( )
午前
・標本整理
午後
・データ解析
・論文書き(ワラジムシ群集)
種の分布解析でロジスティック解析をする際、対象種が確認された在データだけでなく、調べたが対象種が確認できなかった不在データも必要となる。
私の場合、採集日や場所を紐付けした動物標本のデータと、別に、調査を行なった地点のデータを記録している。
したがって、ロジスティック解析をするには、不在データを準備して、標本データに結合する必要がある。
これまで手作業でやっていたのだが、「dplyr」パッケージの「anti_join( )」を使うことでどうにかできた。流れとしては、調査地点データから「標本データに含まれている地点の行を削除」したのち、これら2つのデータを結合した。
標本データ。「species.csv」として記録されているとする。
地点データ。「plot.csv」として記録されているとする。
データの取り込みと、2つのデータの列が異なるので揃える。
「spe」において、1地点で複数のサンプルが記録されており、1個体/1種/1地点、する場合は、「dplyr::distinct( )」や「duplicated( )」で削除できるはずだが、複数の列を対象とする場合は試したことがないので、良く分からない。
・標本整理
午後
・データ解析
・論文書き(ワラジムシ群集)
種の分布解析でロジスティック解析をする際、対象種が確認された在データだけでなく、調べたが対象種が確認できなかった不在データも必要となる。
私の場合、採集日や場所を紐付けした動物標本のデータと、別に、調査を行なった地点のデータを記録している。
したがって、ロジスティック解析をするには、不在データを準備して、標本データに結合する必要がある。
これまで手作業でやっていたのだが、「dplyr」パッケージの「anti_join( )」を使うことでどうにかできた。流れとしては、調査地点データから「標本データに含まれている地点の行を削除」したのち、これら2つのデータを結合した。
標本データ。「species.csv」として記録されているとする。
地点データ。「plot.csv」として記録されているとする。
データの取り込みと、2つのデータの列が異なるので揃える。
spe <- read.csv("species.csv", header=T)
plot <- read.csv("plot.csv", header=T)
#種データの「spe」列を作成し「NONE」と記入する
plot$spe <- rep("NONE", nrow(plot))
plot <- plot[, c(3,1,2,4)] #列の順番を「spe」データと揃える
標本データ「spe」に含まれる地点 (site列) を全調査地点データ「plot」の地点 (site列) と比較し、同じ地点名がある場合は「plot」からその地点名がある行を削除する。削除した後の「plot」のデータは「absent」に格納する。
#install.packages("dplyr", repos="http://cran.ism.ac.jp/")
library(dplyr)
absent <- anti_join(plot, spe, by = "site")
「spe」と「absent」を結合する。
library(dplyr) pre_ab <- bind_rows(spe, absent)「sort( )」しているが、下記のように「Burmoniscus ocellatus」がいた地点と、本種が確認されなかった地点「NONE」が混じったデータが出来上がった。
「spe」において、1地点で複数のサンプルが記録されており、1個体/1種/1地点、する場合は、「dplyr::distinct( )」や「duplicated( )」で削除できるはずだが、複数の列を対象とする場合は試したことがないので、良く分からない。
2020年3月27日金曜日
葉面積の測定
午前
・標本整理
午後
・データ解析
総説を書く必要があり、次いでに昔のデータも解析して載せちゃおうと考えている。
10年以上前、まだ、西表島の住民だった頃に行った、Burmoniscus ocellatusによる4樹種の落葉の摂食実験。
日毎の摂食量は葉面積で評価する。したがって、葉面積の測定を行う必要がある。ImageJとか専用のソフトとか色々な方法があるのだが、せっかくなのでRで、と考えたのが間違いだった?
すぐに、「LeafArea」パッケージという便利そうなものを発見した。
使い方は簡単で、こんな感じの画像をデスクトップの「data」というフォルダに入れたとする。画像はjpegかtiffでなければならない。ちなみに、jpegの拡張子がjpgだと警告が出た。
下記を実行すればよい。
ただ、それぞれ独立した図形の面積は出力されるのだが、各数値がどの図形を指しているのかが分からない。
色々と悩んだあげく、ImageJを動かして分かったのだが、結果 (「res」の中身) ではなく、解析の途中で出力される下記の「XStart」と「YStart」が左上からの座標を示しているッポイ、ということがやっと分かった。
ただ、これはデータとして出力されない (?) ので、コピペで残すしかなく、それぞれ対応づけをする必要もあるので、あまり良い方法ではない。合計の面積を調べるものみたい。
・標本整理
午後
・データ解析
総説を書く必要があり、次いでに昔のデータも解析して載せちゃおうと考えている。
10年以上前、まだ、西表島の住民だった頃に行った、Burmoniscus ocellatusによる4樹種の落葉の摂食実験。
日毎の摂食量は葉面積で評価する。したがって、葉面積の測定を行う必要がある。ImageJとか専用のソフトとか色々な方法があるのだが、せっかくなのでRで、と考えたのが間違いだった?
すぐに、「LeafArea」パッケージという便利そうなものを発見した。
使い方は簡単で、こんな感じの画像をデスクトップの「data」というフォルダに入れたとする。画像はjpegかtiffでなければならない。ちなみに、jpegの拡張子がjpgだと警告が出た。
下記を実行すればよい。
#install.packages("LeafArea", repos="http://cran.ism.ac.jp/")
library(LeafArea)
ex.dir <- eximg()
res <- run.ij(set.directory = "~/Desktop/data//", #winだと少し違う
distance.pixel = 2480, #画像の幅に含まれるpixel数
known.distance = 21, #画像の幅のcm
save.image = TRUE, #2値化を残すか
log=TRUE) #ログを残すか
res
このデータの黒色の pixel 数から面積が計算される。
ただ、それぞれ独立した図形の面積は出力されるのだが、各数値がどの図形を指しているのかが分からない。
色々と悩んだあげく、ImageJを動かして分かったのだが、結果 (「res」の中身) ではなく、解析の途中で出力される下記の「XStart」と「YStart」が左上からの座標を示しているッポイ、ということがやっと分かった。
ただ、これはデータとして出力されない (?) ので、コピペで残すしかなく、それぞれ対応づけをする必要もあるので、あまり良い方法ではない。合計の面積を調べるものみたい。
2020年3月26日木曜日
OTU名の変更
午前
・修論手伝(標本整理)
・標本整理
午後
・データ解析
ggtreeを使った系統樹描画。OTU名の変更も簡単だった。昨日、うまく行かなかったのは、単純なデータの勘違いだった。
流れは、下記方法で「nwk」データを「tree」に読み込んだ場合、OTU名は「tree$tip.label」に入っているので、「tree$tip.label <- ***」で好きな名前に変更すれば良い。
なぜ、そんなことをする必要があったのかというと、KAKUSAN4を使って同じ配列データを一つのハプロタイプにまとめたので、サンプル数が多いハプロタイプは名前が非常に長かったため。例えば、こんなのとか。
"Amami-38_St-6_109_Amami-38_St-6_110_Uke-1_St-6_119_Uke-1_St-6_120_Uke-1_St-6_121_Miyazaki-1_St-6_132_Amami-6_St-6_160_Amami-6_St-6_163"
これは、各シーケンスデータに「locality_haplotptype id_sample id」が書かれていて、それが9サンプルが合わさったため。
でも、結果オーライなのだが、"_"で区切った2番目にはハプロタイプ番号(St-6)が記されているので、それをOTUに使えばOK!
テキストの一部を切り出すのは「library(stringr)」の「str_split( )」で出来る!
あとは、グループごとに色付けして出力。
・修論手伝(標本整理)
・標本整理
午後
・データ解析
ggtreeを使った系統樹描画。OTU名の変更も簡単だった。昨日、うまく行かなかったのは、単純なデータの勘違いだった。
流れは、下記方法で「nwk」データを「tree」に読み込んだ場合、OTU名は「tree$tip.label」に入っているので、「tree$tip.label <- ***」で好きな名前に変更すれば良い。
なぜ、そんなことをする必要があったのかというと、KAKUSAN4を使って同じ配列データを一つのハプロタイプにまとめたので、サンプル数が多いハプロタイプは名前が非常に長かったため。例えば、こんなのとか。
"Amami-38_St-6_109_Amami-38_St-6_110_Uke-1_St-6_119_Uke-1_St-6_120_Uke-1_St-6_121_Miyazaki-1_St-6_132_Amami-6_St-6_160_Amami-6_St-6_163"
これは、各シーケンスデータに「locality_haplotptype id_sample id」が書かれていて、それが9サンプルが合わさったため。
でも、結果オーライなのだが、"_"で区切った2番目にはハプロタイプ番号(St-6)が記されているので、それをOTUに使えばOK!
テキストの一部を切り出すのは「library(stringr)」の「str_split( )」で出来る!
library(ggtree)
library(treeio)
# nwkデータの取り込み
tree <- read.newick("ML_stimpsonii.nwk")
# ラベル (OTU名) の確認
tree$tip.label
長い、、、
# 上記のデータを対象に、"_"で区切り、その2番目を「tree$tip.label」に入れる library(stringr) # "_"ごとに区切って2番目を取り出す a<-str_split(tree$tip.label, pattern = "_", simplify = TRUE) tree$tip.label <- a[,2] # ラベル (OTU名) の確認 tree$tip.label短くなった。
あとは、グループごとに色付けして出力。
# OTU名に基づきグループを作成する
cls <- list(Kumejima=c("St-55", "St-56"), Okinawa=c("St-57", "St-58"・・・))
# nwkデータにグループ名を紐つけ
tree <- groupOTU(tree, cls)
# 描画
library("colorspace") #色に必要?
ggtree(tree, aes(color=group), layout='circular') +
scale_color_manual(values=c("black", "azure4", "red", "magenta", "royal blue", "#00552e")) +
geom_tiplab()
2020年3月25日水曜日
ggtree
午前
・標本整理
午後
・データ解析
系統樹の描画はこれまで、FigTree + お絵かきソフト、を使っていた。
お絵かきソフトを使うと細かな描画が楽だけど、一斉に指定できないなどの不便がある。
何か良い方法がないかと探していたら、Rのggtreeパッケージを発見した。
ggtreeを使ってRで系統樹を扱う
ggtree: Elegant Graphics for Phylogenetic Tree Visualization and Annotation
Phylogenetic trees in R using ggtree
描画すること自体は簡単。
ただ、OTU名を変更してグループ化したら動かない、、、もう少し勉強が必要。
・標本整理
午後
・データ解析
系統樹の描画はこれまで、FigTree + お絵かきソフト、を使っていた。
お絵かきソフトを使うと細かな描画が楽だけど、一斉に指定できないなどの不便がある。
何か良い方法がないかと探していたら、Rのggtreeパッケージを発見した。
描画すること自体は簡単。
ただ、OTU名を変更してグループ化したら動かない、、、もう少し勉強が必要。
2020年3月24日火曜日
2020年3月23日月曜日
rarefaction curve
午前
・標本整理
午後
・データ解析
・論文書き(ワラジムシ群集)
先週の月曜日の続きで、福岡時代のワラジムシ群集の論文化をすすめる。
4つの環境(人工環境、草地、竹林、森林)で群集比較をする。
個体数、種組成は先週作成したので、本日は、種数の比較。rarefaction curveの出番か。
個体数ベースが良いのか、地点ベースが良いのか悩むが、地点ベースで作図してみた。
「vegan」パッケージのspecaccum()を使えば簡単に描ける。
でも、iNEXT()の方がカッコいい、、、使いこなすのは難しそう。
・標本整理
午後
・データ解析
・論文書き(ワラジムシ群集)
先週の月曜日の続きで、福岡時代のワラジムシ群集の論文化をすすめる。
4つの環境(人工環境、草地、竹林、森林)で群集比較をする。
個体数、種組成は先週作成したので、本日は、種数の比較。rarefaction curveの出番か。
個体数ベースが良いのか、地点ベースが良いのか悩むが、地点ベースで作図してみた。
「vegan」パッケージのspecaccum()を使えば簡単に描ける。
でも、iNEXT()の方がカッコいい、、、使いこなすのは難しそう。
2020年3月18日水曜日
期待
午前
・標本整理
午後 ・標本整理
・データ解析
共同研究者へ送る標本の整理が終わった。
本日は、卒業式。
他の大学と同じように、全体の式はなく証書の授与式のみだった。集合写真もダメだったみたいなので可哀想な気もするが、「コロナの時の、、、」といつまでも記憶になることは間違いない。
研究室からは4名(+1になる予定)の学生が旅立った。鳥取大学では普通なのだが、全員、鳥取県外の学生なので鳥取を離れることになる。
福岡時代は、教師になる人が多かったこともあるが、卒業後、多くが福岡に住んでいたのとは大きな違いである。
私が鳥取に来たのが4年前なので、今年の卒業生と鳥取で同じ時間を過ごしたことになる。ありきたりだけど、長いようであっという間の4年間だった。
鳥取に来た、ということもないだろうが(年をとったため?)、個人ゼミとか、Rで作図指導とか、学生への接し方が少し変わったかな。それが効を奏したのか、今年の卒論はかなりスムーズだった気がする。むしろ、その余裕で12〜1月にjavaとかphpとかにハマってしまい、時間的な余裕はなかったが。
「何が起こっても不思議ではない時代」を痛感する日々。大変なことも多いと思うけど、きっと元気にやっていける、、、と期待している!
・標本整理
午後 ・標本整理
・データ解析
共同研究者へ送る標本の整理が終わった。
本日は、卒業式。
他の大学と同じように、全体の式はなく証書の授与式のみだった。集合写真もダメだったみたいなので可哀想な気もするが、「コロナの時の、、、」といつまでも記憶になることは間違いない。
研究室からは4名(+1になる予定)の学生が旅立った。鳥取大学では普通なのだが、全員、鳥取県外の学生なので鳥取を離れることになる。
福岡時代は、教師になる人が多かったこともあるが、卒業後、多くが福岡に住んでいたのとは大きな違いである。
私が鳥取に来たのが4年前なので、今年の卒業生と鳥取で同じ時間を過ごしたことになる。ありきたりだけど、長いようであっという間の4年間だった。
鳥取に来た、ということもないだろうが(年をとったため?)、個人ゼミとか、Rで作図指導とか、学生への接し方が少し変わったかな。それが効を奏したのか、今年の卒論はかなりスムーズだった気がする。むしろ、その余裕で12〜1月にjavaとかphpとかにハマってしまい、時間的な余裕はなかったが。
「何が起こっても不思議ではない時代」を痛感する日々。大変なことも多いと思うけど、きっと元気にやっていける、、、と期待している!
2020年3月17日火曜日
カーネル密度推定
午前
・標本整理
午後
・データ解析
・論文書き(マダニ卒論論文化)
今日はマダニ卒論の論文化を頑張るつもりだったのだが、最近、開始した共同研究のデータ解析について相談があり、それに時間がかかってしまった。まだ、回答できていないが。
論文化は、文章書きをやるつもりだったが、調子が狂ったという言い訳で作図に手を出してしまった。
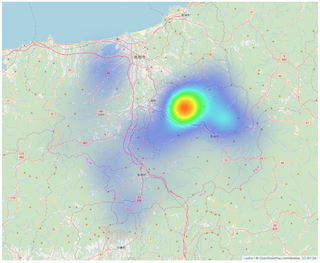
leafletでは、leaflet.extrasパッケージのaddHeatmap() を使うと、ヒートマップを簡単に作ることができる。
密度を色で表現していることは良く分かるのだが、その原理がよく分からない。
で、同じような?方法として、むしろ一般的なのはカーネル密度推定。RでもGISにカーネル密度推定を利用する方法は色々とあるみたいなのだが、leafletに掲載する方法はあまり見当たらない。
非常に分かりやすいサイトを発見したので、早速試してみた。図は作れたが、、、詳細を理解できない。
予想よりも密度が非常に低いのだが、これはグリッドサイズの問題?地点数が少ないから?
・標本整理
午後
・データ解析
・論文書き(マダニ卒論論文化)
今日はマダニ卒論の論文化を頑張るつもりだったのだが、最近、開始した共同研究のデータ解析について相談があり、それに時間がかかってしまった。まだ、回答できていないが。
論文化は、文章書きをやるつもりだったが、調子が狂ったという言い訳で作図に手を出してしまった。
leafletでは、leaflet.extrasパッケージのaddHeatmap() を使うと、ヒートマップを簡単に作ることができる。
密度を色で表現していることは良く分かるのだが、その原理がよく分からない。
で、同じような?方法として、むしろ一般的なのはカーネル密度推定。RでもGISにカーネル密度推定を利用する方法は色々とあるみたいなのだが、leafletに掲載する方法はあまり見当たらない。
非常に分かりやすいサイトを発見したので、早速試してみた。図は作れたが、、、詳細を理解できない。
予想よりも密度が非常に低いのだが、これはグリッドサイズの問題?地点数が少ないから?
2020年3月16日月曜日
価値はある
午前
・標本整理
午後
・論文書き(ワラジムシ群集)
随分前(福岡時代)に採集したデータの論文化。
人工環境、草地、竹林、森林でワラジムシ群集を比較した、という良くあるテーマで結果も予想がつくのだが、実はその手の研究で(日本のデータで)引用できるものがあまり無い。また、外来種竹林の影響を評価している点で、一応、論文にする価値はあるだろう。
調査地点は、合計で102地点、採集個体数は約1万。
図の作成は終わった。leafletとplotで色が少し違って見えるので、このあたりの修正は必要そうだ。
地図に時間をかけなくて良くなったのが、ここ最近の成長か。
結果はありきたり。
久しぶりに座標付けもしてみた。「R NMDS」で検索したら、自分で書いた記事がヒットした。
指標種分析というのをやってみた。とりあえず結果は出たが、解析過程が全く分からない、、、。在来種が撹乱環境の指標種になっているのは興味深いかも?
・標本整理
午後
・論文書き(ワラジムシ群集)
随分前(福岡時代)に採集したデータの論文化。
人工環境、草地、竹林、森林でワラジムシ群集を比較した、という良くあるテーマで結果も予想がつくのだが、実はその手の研究で(日本のデータで)引用できるものがあまり無い。また、外来種竹林の影響を評価している点で、一応、論文にする価値はあるだろう。
調査地点は、合計で102地点、採集個体数は約1万。
図の作成は終わった。leafletとplotで色が少し違って見えるので、このあたりの修正は必要そうだ。
地図に時間をかけなくて良くなったのが、ここ最近の成長か。
結果はありきたり。
久しぶりに座標付けもしてみた。「R NMDS」で検索したら、自分で書いた記事がヒットした。
指標種分析というのをやってみた。とりあえず結果は出たが、解析過程が全く分からない、、、。在来種が撹乱環境の指標種になっているのは興味深いかも?
2020年3月13日金曜日
フサヤスデ
午前
・標本整理
午後
・データ整理
・メール書き
共同研究者に送る標本整理、、、終わらない。
放ったらかしにしたツケが。
Karasawa S., Kawano K., Fukaya S., Tsurusaki N. (2020)
Upgrading of three subspecies of Eudigraphis takakuwai to the species rank (Diplopoda: Penicillata: Polyxenida: Polyxenidae). Species Diversity, 25:89–102
先日、アクセプトされた論文がweb公開されていた。
フサヤスデの学名に関する論文です。これまで亜種として扱われていたものを、分布や遺伝子データから種に格上げしました。
遺伝子解析の部分を担当しました。
・標本整理
午後
・データ整理
・メール書き
共同研究者に送る標本整理、、、終わらない。
放ったらかしにしたツケが。
Karasawa S., Kawano K., Fukaya S., Tsurusaki N. (2020)
Upgrading of three subspecies of Eudigraphis takakuwai to the species rank (Diplopoda: Penicillata: Polyxenida: Polyxenidae). Species Diversity, 25:89–102
先日、アクセプトされた論文がweb公開されていた。
フサヤスデの学名に関する論文です。これまで亜種として扱われていたものを、分布や遺伝子データから種に格上げしました。
遺伝子解析の部分を担当しました。
2020年3月12日木曜日
地点ごとの外来種・在来種ごとの種数を算出
午前
・標本整理
午後
・授業準備
先日の続きで、新しいカテゴリーとして、外来種と在来種を加えて、地点ごとの外来種・在来種ごとの種数を算出する方法。
一発でやる方法は分からず、まどろっこしい方法でどうにかできた。
元データはこんな感じ。
まず、ここに外来種と在来種の情報を加える。
流れとしては、1)種名が書かれている「spe」をコピーして、新しい列「origin」として貼り付ける。そして、2)各種名ごとに外来種(exotic) or 在来種(native)に置換する。
次に、外来種・在来種ごと「origin」に、地点ごと「spe」に種数「n_spe」を算出する。これは先日の方法と同じ。
で、現在の縦長のデータ(スタック形式)を横長の(アンスタック形式)にすると、欲しかったものが手にはいる。
このように、地点「site」ごとに外来種(exotix)と在来種(native)の種数が算出されている。
・標本整理
午後
・授業準備
先日の続きで、新しいカテゴリーとして、外来種と在来種を加えて、地点ごとの外来種・在来種ごとの種数を算出する方法。
一発でやる方法は分からず、まどろっこしい方法でどうにかできた。
元データはこんな感じ。
まず、ここに外来種と在来種の情報を加える。
流れとしては、1)種名が書かれている「spe」をコピーして、新しい列「origin」として貼り付ける。そして、2)各種名ごとに外来種(exotic) or 在来種(native)に置換する。
#データが「data_exu」に入ってるとして
#install.packages("tidyverse", repos="http://cran.ism.ac.jp/")
library(tidyverse) #文字変換に必要
data_exu$origin <- data_exu$spe #「origin」を作成し、そこに「spe」の情報を入れる
data_exu$origin <- data_exu$origin
%>% str_replace_all(c("Agnara pannuosa"="native",
・・・ "Armadillidium vulgare"="exotic",・・・)) #"置換前"="置換後"
次に、外来種・在来種ごと「origin」に、地点ごと「spe」に種数「n_spe」を算出する。これは先日の方法と同じ。
library(dplyr)
spe_site2 <- data_exu %>% group_by(origin, site)
%>% summarize(n_spe = n_distinct(spe)) #siteごとに分けて,speのユニークな数.
spe_site2 <- data.frame(spe_site2)
で、現在の縦長のデータ(スタック形式)を横長の(アンスタック形式)にすると、欲しかったものが手にはいる。
library(tidyverse) #pivot_widerに必要
spe_site3 <- data.frame(spe_site2
%>% pivot_wider(names_from = origin, values_from = n_spe))
上記は「origin」を列名にすると解釈すれば良い。
このように、地点「site」ごとに外来種(exotix)と在来種(native)の種数が算出されている。
2020年3月11日水曜日
2020年3月10日火曜日
地点あたりの種数算出
午前
・標本整理
午後
・論文書き(卒論論文化: マダニ)
昨日、悩んだ地点あたりの種数算出は、家に帰って考えたらアッサリ解決した。
データはこんな感じで、1行1個体で「site」列に地点、「spe」列に種名が書かれている。
例えば、地点ごとの個体数を算出するには下記でOK!
これは「site」ごとに分割して、「length」でその分割された行の数を数えていると考えると良い。
で、この方法を応用して、「site」ごとに分割して「nlevels」で分割されたデータの「spe」に含まれるFactor(種名)の数を数えれば良いと考えたのが昨日。
「factor()」とか試したけどダメ!
で、たどり着いたのが、「library(dplyr)」の「group_by」+「n_distinct」。
「group_by」は文字通りグループに分ける。で、「n_distinct」は「ユニーク数を数える(重複を除去して数える)」という機能。正にコレが欲しかった!
さらに、元データに外来種と在来種の情報を加えて、地点ごとの外来種と在来種の種数を調べたのが、これはさらに少し手を加える必要があった。
・標本整理
午後
・論文書き(卒論論文化: マダニ)
昨日、悩んだ地点あたりの種数算出は、家に帰って考えたらアッサリ解決した。
データはこんな感じで、1行1個体で「site」列に地点、「spe」列に種名が書かれている。
例えば、地点ごとの個体数を算出するには下記でOK!
#データが「data_exu」に入ってるとして tapply(data_exu$spe, data_exu$site, length)場所ごとの種ごとの個体数ならば
tapply(data_exu$spe, list(data_exu$site, data_exu$spe), length)で良い。
これは「site」ごとに分割して、「length」でその分割された行の数を数えていると考えると良い。
で、この方法を応用して、「site」ごとに分割して「nlevels」で分割されたデータの「spe」に含まれるFactor(種名)の数を数えれば良いと考えたのが昨日。
tapply(data_exu$spe, data_exu$site, nlevels)なぜか、全て16になってしまう。16はデータに含まれる種数。
「factor()」とか試したけどダメ!
で、たどり着いたのが、「library(dplyr)」の「group_by」+「n_distinct」。
「group_by」は文字通りグループに分ける。で、「n_distinct」は「ユニーク数を数える(重複を除去して数える)」という機能。正にコレが欲しかった!
library(dplyr) a <- data_exu %>% group_by(site) %>% summarize(n_spe = n_distinct(spe)) aで良い、、、と思う。「group_by(site)」で「site」ごとに分けて、「spe」に含まれるユニーク数(種数)を数えて「n_spe」に返してくれる。
さらに、元データに外来種と在来種の情報を加えて、地点ごとの外来種と在来種の種数を調べたのが、これはさらに少し手を加える必要があった。
2020年3月9日月曜日
2020年3月6日金曜日
2020年3月5日木曜日
2020年3月4日水曜日
2020年3月3日火曜日
Rmarkdown
午前
・標本整理
午後
・授業準備
授業の準備でハマって半日が終わった。
Rmarkdownで作成したドキュメントをweb経由で学生に配布することを考えた。
htmlが出力されているのだから、そのままサーバーに載せたらOK、と思ったそうはいかず。
ネットで調べたらGitHubが常套手段のようだが、GitHubが扱ったことなく、悪戦苦闘の末、、、できない。
このサイトによると、「_site.yml」と「.Rmd」をおいて「rmarkdown::render_site()」を実行して出力されたファイルをサーバーに載せれば良いみたい。
どうにかできた。そのうち、GitHubに移行したい。
・標本整理
午後
・授業準備
授業の準備でハマって半日が終わった。
Rmarkdownで作成したドキュメントをweb経由で学生に配布することを考えた。
htmlが出力されているのだから、そのままサーバーに載せたらOK、と思ったそうはいかず。
ネットで調べたらGitHubが常套手段のようだが、GitHubが扱ったことなく、悪戦苦闘の末、、、できない。
このサイトによると、「_site.yml」と「.Rmd」をおいて「rmarkdown::render_site()」を実行して出力されたファイルをサーバーに載せれば良いみたい。
どうにかできた。そのうち、GitHubに移行したい。
2020年3月2日月曜日
生態学は環境問題を解決できるか?
午前
・標本整理
午後
・会議
・データ整理


生態学は環境問題を解決できるか? (共立スマートセレクション)
授業で少しは触れていていたが、上手く話すことができなかったことがスッキリした。本書のフレーズを4月からの授業で何度も話すだろう。
正直なところ、タイトルを見て、生態学的な観点から環境の現状を示した本を想像し、あまり興味が惹かれず購入しようか迷った。買って大正解。
著者の専門は、植物生態学、ビックデータ解析、だそうで、特に、コンピュータを駆使して、シミュレーションやAIによる生物分布調査などを行なっている。
しかし、本書の半分以上は、著者の研究の紹介よりも、環境倫理や著者の経験に基づいた環境問題に対峙する私たちのあるべき姿勢に関する話題に割かれている。
著者曰く、環境問題で大事なことは「答えがないことを知ることが答え」だ!
これだけではよく分からない。すなわち、環境問題においては、「絶対的な答えがある」という信念は捨て、その時に応じて、最も良い答えを導き出すことこそが重要なのだ。
「Yes or No」、「All or Nothing」、「白か黒か」ではなく、曖昧な姿勢が重要!でも、実は、これがとても難しい。
例えば、外来種は駆除すべきなのか?
在来の生態系に甚大な被害を及ぼす外来種は、やはり駆除すべきだろう。では、日本中に生息している外来種オカダンゴムシを何千億円も使って駆除する価値はあるだろうか?
また、農作物の多くは外来種である。これらは栽培してはいけないのか。「絶対的な答えはなく」その都度、様々なことを踏まえて答えを見つけるしかないのである。
環境倫理の考えの中で、「shallow ecology」、「deep ecology」という考えがあるそうだ。前者は、「人間にとって有用だから自然を守る」という考えで、後者は、「自然は存在そのものに価値があるため、全てを守る必要がある」という考え。
後者は素晴らしい考えだが、極論的には全ての文明を捨てて原始の生活を送る必要があり、実行は非現実的だろう。
前者は、いわゆる生態系サービスであり、自然の保護をする上で広く受け入られやすい。しかし、この考えでは、価値が低いとレッテルを貼られた自然は壊されてしまう(利用されていない価値を評価することもできるが)。本当にそれで良いのだろうか。そもそも、その価値評価が人によってずれることもある。
やはり、「どちらが良い」とは決めることができず、その都度、どちらの立場に立つか判断することが現実的だろう。
「その都度、、、」と書くと何か弱腰のように思えるが、「optimistic pessimist(楽観的悲観主義者)」という言葉を知ると、その重要性が理解できる。
「楽観的」で「悲観的」とは、まず、現在、生じている環境問題(温暖化とか、マイクロプラスチックとか、外来種とか)の悲惨さについては深刻に受け止める。この点では、悲観的にあるべきだ。
しかし、ここで、「も〜だめだ」と諦めるのではなく、「やれることをやろう」と前向きに楽観的になることが大事なのだ。納得だ!
「Yes or No」の立場で極論に突っ走ることもできるかも知れないが、現実的にできる選択肢の中から最も良いものを選ぶ姿勢こそが、「やれることをやろう」につながると思う。
「答えがない」ことに、「より良い答え(やれること)を見つける」ために知識を蓄える場所、大学はそいう所だと思い出した、、、!
また、「NIMBY」、「technological optimism」、「VHEMT」など初耳の言葉も多くあり、環境倫理を勉強する必要があると痛感した。
大学生には是非、一読して、テスト勉強ではない勉強をしなければ、そんなことを考えて欲しい。必読の一冊。
アメリカの大学に進学した理由など、自伝的記述も面白い。
もくじ
1.人と自然と環境問題
2.環境倫理と歴史
3.答えはひとつにきまらない
4.外来種のおはなし
5.前向きになんとかしよう
6.科学者とは・科学とは
7.全力で走らねば
・標本整理
午後
・会議
・データ整理

生態学は環境問題を解決できるか? (共立スマートセレクション)
授業で少しは触れていていたが、上手く話すことができなかったことがスッキリした。本書のフレーズを4月からの授業で何度も話すだろう。
正直なところ、タイトルを見て、生態学的な観点から環境の現状を示した本を想像し、あまり興味が惹かれず購入しようか迷った。買って大正解。
著者の専門は、植物生態学、ビックデータ解析、だそうで、特に、コンピュータを駆使して、シミュレーションやAIによる生物分布調査などを行なっている。
しかし、本書の半分以上は、著者の研究の紹介よりも、環境倫理や著者の経験に基づいた環境問題に対峙する私たちのあるべき姿勢に関する話題に割かれている。
著者曰く、環境問題で大事なことは「答えがないことを知ることが答え」だ!
これだけではよく分からない。すなわち、環境問題においては、「絶対的な答えがある」という信念は捨て、その時に応じて、最も良い答えを導き出すことこそが重要なのだ。
「Yes or No」、「All or Nothing」、「白か黒か」ではなく、曖昧な姿勢が重要!でも、実は、これがとても難しい。
例えば、外来種は駆除すべきなのか?
在来の生態系に甚大な被害を及ぼす外来種は、やはり駆除すべきだろう。では、日本中に生息している外来種オカダンゴムシを何千億円も使って駆除する価値はあるだろうか?
また、農作物の多くは外来種である。これらは栽培してはいけないのか。「絶対的な答えはなく」その都度、様々なことを踏まえて答えを見つけるしかないのである。
環境倫理の考えの中で、「shallow ecology」、「deep ecology」という考えがあるそうだ。前者は、「人間にとって有用だから自然を守る」という考えで、後者は、「自然は存在そのものに価値があるため、全てを守る必要がある」という考え。
後者は素晴らしい考えだが、極論的には全ての文明を捨てて原始の生活を送る必要があり、実行は非現実的だろう。
前者は、いわゆる生態系サービスであり、自然の保護をする上で広く受け入られやすい。しかし、この考えでは、価値が低いとレッテルを貼られた自然は壊されてしまう(利用されていない価値を評価することもできるが)。本当にそれで良いのだろうか。そもそも、その価値評価が人によってずれることもある。
やはり、「どちらが良い」とは決めることができず、その都度、どちらの立場に立つか判断することが現実的だろう。
「その都度、、、」と書くと何か弱腰のように思えるが、「optimistic pessimist(楽観的悲観主義者)」という言葉を知ると、その重要性が理解できる。
「楽観的」で「悲観的」とは、まず、現在、生じている環境問題(温暖化とか、マイクロプラスチックとか、外来種とか)の悲惨さについては深刻に受け止める。この点では、悲観的にあるべきだ。
しかし、ここで、「も〜だめだ」と諦めるのではなく、「やれることをやろう」と前向きに楽観的になることが大事なのだ。納得だ!
「Yes or No」の立場で極論に突っ走ることもできるかも知れないが、現実的にできる選択肢の中から最も良いものを選ぶ姿勢こそが、「やれることをやろう」につながると思う。
「答えがない」ことに、「より良い答え(やれること)を見つける」ために知識を蓄える場所、大学はそいう所だと思い出した、、、!
また、「NIMBY」、「technological optimism」、「VHEMT」など初耳の言葉も多くあり、環境倫理を勉強する必要があると痛感した。
大学生には是非、一読して、テスト勉強ではない勉強をしなければ、そんなことを考えて欲しい。必読の一冊。
アメリカの大学に進学した理由など、自伝的記述も面白い。
もくじ
1.人と自然と環境問題
2.環境倫理と歴史
3.答えはひとつにきまらない
4.外来種のおはなし
5.前向きになんとかしよう
6.科学者とは・科学とは
7.全力で走らねば
登録:
投稿 (Atom)